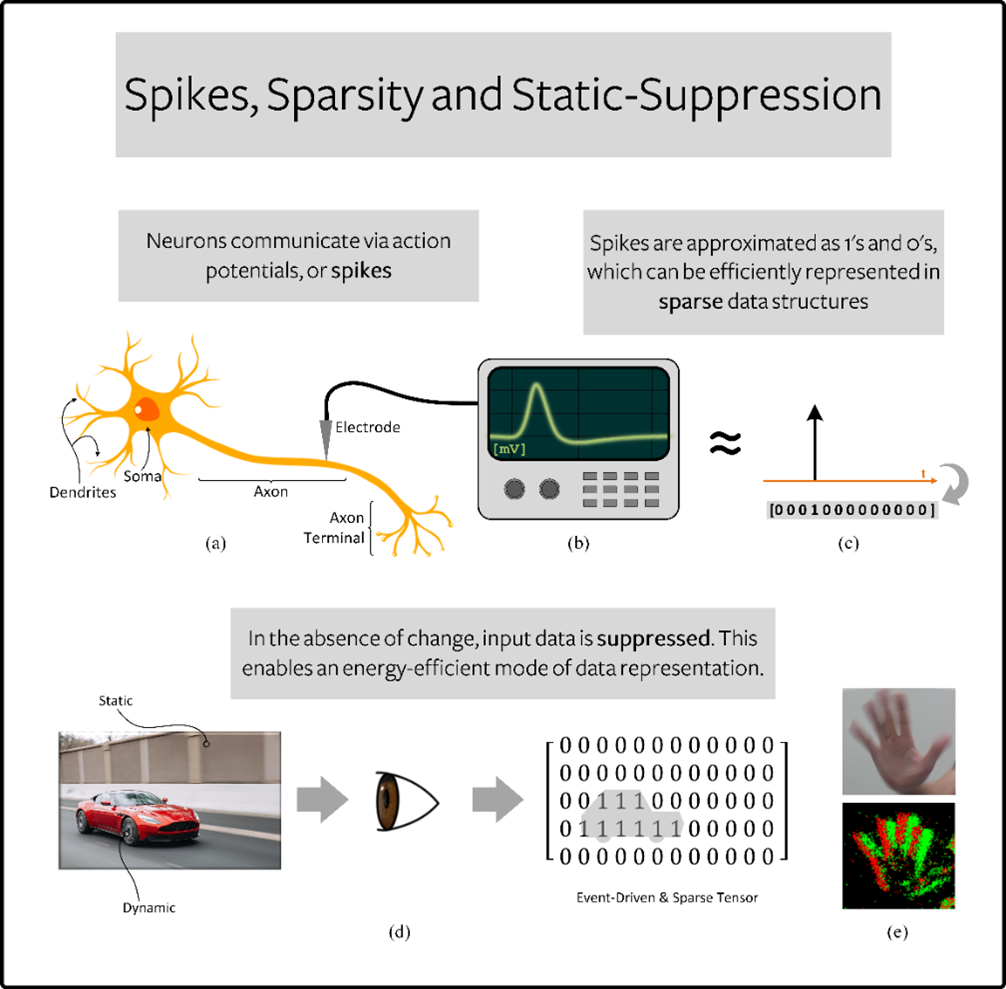
Nach all dem Tagesgeschäft, das uns in der Folge unserer Jahreskonferenz Scion schnell wieder ereilte, nehmen wir einen Rückblick auf Scion Herbst 2018 vor. Ein ganz großer Dank geht an unsere Partner und insbesondere an die Speaker, Teilnehmerinnen und Teilnehmer, ohne die Scion 2018 in dieser Größenordnung und mit einem solch positiven Widerhall überhaupt nicht möglich gewesen wäre. Nicht für umsonst haben wir dem Veranstaltungsformat den Namen „Scion“ gegeben. Es handelt sich um einen Sprössling, der gute Rahmenbedingungen und Zeit zum Wachsen braucht, genau wie das Thema „Vertrauen in KI“ in der Medizinforschung. Wir freuen uns, dass wir die interdisziplinäre Wissensvermittlung und den Dialog zum Thema Künstliche Intelligenz in Life Science eröffnen und intensivieren konnten. Eure aktive Mitwirkung, hochqualitative Vorträge, spannende Diskussionen und das positive Feedback bestätigen unsere Intention, das Vertrauen in die Künstliche Intelligenz weiterhin zu etablieren.
Die Social Media Wall, die wir bei unserer Veranstaltung auf einem großen Bildschirm projiziert hatten, machte unsere Mission unter dem Hashtag #Scion2018 für alle Teilnehmenden vor Ort und über die meisten sozialen Netzwerke erlebbar. Für einen fotografischen Rückblick bitte hier entlang: Fotos Scion Herbst 2018.
Nachfolgend seien Moderatoren und Speaker genannt, die jeweils auf ihren Fachgebieten mit qualitativ hochwertigen Vorträgen und Beiträgen Scion Herbst 2018 zu einem besonderen Erlebnis für alle Teilnehmenden werden ließen. Die Eröffnung und Moderation wurden übernommen von Nathalie Biniaminov, während Prof. Thomas Hirth (beide KIT) Scion eröffnete und seitens des KIT ein Grußwort an die Teilnehmerinnen und Teilnehmer entsendete. Inbegriffen war eine Vorstellung des KIT, die u.a. die interdisziplinären Lehrgebiete und Forschungsfelder aus der Symbiose der Uni Karlsruhe und des Forschungszentrums Karlsruhe vermittelte.

Nathalie Biniaminov – Akademische Mitarbeiterin am IfSS KIT

Nathalie Biniaminov, Thomas Hirth ( beide KIT) Sergey Biniaminov ,Viktor Eberhardt (beide HS Analysis GmbH) (v.l.n.r.)

Prof. Dr. Thomas Hirth – Vizepräsident Innovation und Internationales KIT
Unter den Vortragenden wurden die Themen Diagnostik, Histologie und der Einsatz der KI bei der Datenanalyse repräsentiert durch PD Dr. Afsaneh Soruri (Pathologie Pforzheim), Prof. Thomas Rüdiger (Pathologie Karlsruhe), Prof. Timo Gaiser (Pathologie Mannheim), Dr. Fermin Person und Tim Petschull (Pathologie Hamburg), PD Dr. Gian Kayser (Pathologie Freiburg).
Dr. Soruri hat ihre Expertise in der Diagnostik von Lungenkrebs, die Bedeutung von PD-L1 beim Tumorwachstum und die zellfreie DNA als Biomarker für Krebstherapie vermittelt. Sie hat in ihrem Vortrag die Verbindung zu den Methoden aus der anatomischen, zellulären, molekularen und prädiktiven Pathologie hergestellt.

PD Dr. Afsaneh Soruri – Institut für Pathologie & Molekularpathologie Pforzheim
Während Prof. Rüdiger auf den Prozess in einer Pathologieabteilung eingegangen ist und die Einzelne Schritte von der Probeentnahme bis zum Befund verdeutlichte. Er zeigte an einem Beispiel die Grundlagen in der prädiktiven Pathologie und Klassifikation über die Morphologie und intrinsische Subtypen vom Mammakarzinom. Er verdeutlichte, dass die strukturierte Aufbereitung der Daten eine entscheidende Voraussetzung für den optimalen Wissenstransfer in der evidenzbasierten Medizin spielt.

Prof. Dr. med. Thomas Rüdiger – Leiter der Pathologie am Städtischen Klinikum Karlsruhe
Prof. Gaiser fokussierte sich in seinem Vortrag auf das kolorektale Karzinom und gab einen Überblick zur Korrelation zwischen dem Tumor budding und dem Nodalstatus bzw. der Überlebenswahrscheinlichkeit der Patienten. Er stellte die Ergebnisse der automatischen Detektion in WSI und TMA von Tumorknospen mit konventioneller Segmentierung und mittels anschließender AI-Validierung mit CNNs vor.

Prof. Dr. med. Timo Gaiser – Leitender Oberarzt Pathologie Uniklinik Mannheim
Herr Dr. Person zeigte am Beispiel der Nierenschnitte die Möglichkeiten in der Datenanalyse durch Deep Learning und sprach über die optimale Kombination von Pathologe und Maschine in der Trippeldiagnostik, die auf den histologischen Färbungen, IHC und Elektronenmikroskopie basiert. Während Herr Petschull sein Paper präsentierte, in welchem er durch Proximity Ligation Assay Technik und die modernen Bildanalysemethoden die Interaktion von FHR2 und Laminin Beta 2 bei der Krankheit Glomerulonephritis beschrieben hat.

Dr. Fermin Person – Arzt Nephropathologie UKE Hamburg

Tim Petschull – Doktorand UKE Hamburg
Dr. Kayser verdeutlichte in seinem Vortrag die virtuelle Pathologie, WSI, Integration von LIS, das strukturierte Reporting und computational pathology als wesentliche Bestandteile der digitalen Pathologie. Er beschrieb, wie CNNs und die Kombination der objektbasierten Klassifikation und Texturklassifikation in der modernen Bildanalyse zu den optimalen Ergebnissen in der Diagnostik führen können. So präsentierte er die Ergebnisse der Mitosendetektierung mit DNNs in der Brustkrebsdiagnostik und zeigte die Blackbox von Deep Learning.

PD Dr. Gian Kayser – Leitender Oberarzt Pathologie Uniklinik Freiburg
Zum Thema moderne Methoden in der Medikamentenentwicklung und Therapien sprachen bei Scion Dr. Christoph Schatz (Bayer Pharma AG) und Prof. Alexander Nesterov-Müller (KIT). Beide sehen große Chancen in der Erforschung neuer Medikamente durch die Analyse der Daten mit Deep Learning Verfahren.
Dr. Schatz präsentierte die Untersuchungen zur Wirksamkeit von Antikörper-Therapien bei der Behandlung des Ovarialkarzinoms und stellte die Tumorschrumpfungen nach Bevacizumab Kombinationstherapie vor. Bei seinem Vortrag ging er auf die Quantifizierung der CD31 Gefäßfärbung und Tumor infiltrierenden Immunzellen ein. Als Beispiel für die personalisierte Medizin nahm Dr. Schatz die Quantifzierung der braunen Färbung vom Antigen des Thorium-Konjugat als Target Expression bzw. Target Gehalt in humanen Lungentumoren. Für die Identifikation von Target positiven Patientenpopulationen war es für ihn insbesondere interessant die Häufigkeit und die Konzentration, in der das Target in humanen Tumoren exprimiert ist.

Dr. Christoph Schatz – Senior Scientist, Biomarker Research bei Bayer Pharma AG
Prof. Nesterov-Mueller referierte über das High Throughput Screening mit molekularen Suchmaschinen in der Medikamentenentwicklung. Er verdeutlichte die kombinatorische Peptidsynthese als eine Adressierung bzw. ein Kopplungsschritt für alle Aminosäuren auf einem hochdichten Mikrosstruktur-Piptidarray. Das Ziel ist es, die Nebenwirkungen, die beim Einsatz eines rekombinanten Antikörpers als Medikament auftreten könnten, vorherzusagen. Mit Deep Learning Modellen können die Peptidsequenzen, die für das Binden des Antikörpers verantwortlich sind, den Proteinen, in denen die Sequenz vorkommt, zugeordnet werden können.

Prof. Dr. Alexander Nesterov-Müller – Gruppenleiter am Institute of Microstructure Technology KIT
Das komplexe Feld der Mikroskopie wurde in Theorie und Praxis (Workshops) abgedeckt von Dr. Bernhard Schwarz, Thomas Betz und Detlef Lang (alle Carl Zeiss Microscopy GmbH).
Dr. Schwarz hat in seinem Vortrag eine übersichtliche Einführung in die Zeiss Mikroskopie gegeben und entlang von Kundenanforderungen die Zeiss Digitalisierungsstrategie vorgestellt. Er ging auf einige Bespiellösungen aus dem Bereich korrelative Software, Machine Learning für Segmentierungen und die Plattform APEER zum Austausch von Image Processing Aufgaben ein.
Damit die Scion Teilnehmer vor Ort die Entstehung der digitalen Mikroskopiedaten erleben durften, hat Detlef Lang das digitale Mikroskop ZEISS Smartzoom 5 aufgebaut und in einem Workshop die Methoden zur optischen Abbildung in der Mikroskopie vermittelt. Seinen Schwerpunkt legte er hierbei auf Materialanwendungen sowohl in der Industrie als auch in der akademischen Welt.
Ebenfalls hat Herr Betz einen wertvollen Einblick in die Theorie und Praxis der digitalen Mikroskopiedaten (WSI) gegeben. Scion Teilnehmer durften vor Ort die automatische Mikroskopiestation ZEISS Axio Scan Z.1 erleben und die Digitalisierung der histlogischen Schnitte in einem Workshop begleiten.

Dr. Bernhard Schwarz – Vice President Zeiss Digital Solutions and Strategy

Detlef Lang – Produktspezialist im Vertrieb Zeiss

Thomas Betz – Spezialist WSI Zeiss Mikroskopie Vertrieb

Thomas Betz WSI Workshop am Axio Scan Z.1

Zeiss Axio Scan Z.1

Scion 2018 Zeiss Workshop Teilnehmer
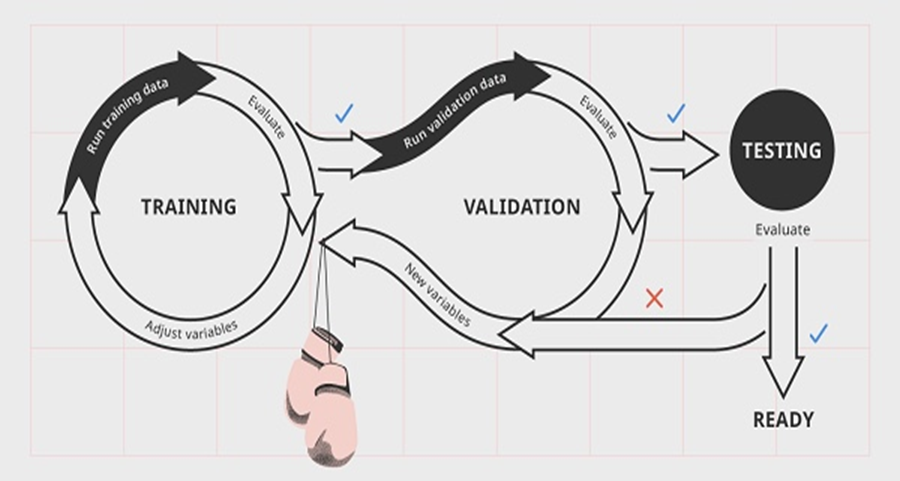
Deep Learning in der Mikroskopie und Bildanalyse war das Hauptthema von Viktor Eberhardt, Prof. Dr. Sascha Seifert und Sergey Biniaminov (vom Team HS Analysis). Die Experten für das Management der Mikroskopiedaten haben Risiken aber auch Chancen kommuniziert, die durch den Einsatz von Deep Learning entstehen können. Anschließend wurden Deep Learning Konzepte wie RetinaNet, YOLO und die intelligente Erstellung der Ground Truth Daten vorgestellt. Das Ziel ist die Entstehung der Schnittstelle zwischen Mensch-Maschine-Kollaboration.
„Für den fortgeschrittenen Einsatz der Deep Learning Modelle im medizinischen Kontext benötigen wir als Data Scientists nicht nur die automatische Optimierung der Modelle, beispielsweise durch die Hypoparameteroptimierung mit Hilfe einer weiteren KI sondern auch Tools für die visuelle Unterstützung der Ergebnisauswertung. Dies macht es möglich die Mensch-Maschine-Kollaboration in der Medizin zu etablieren und somit das Vertrauen in KI unter den Ärzten und Forschern zu steigern.“
Sergey Biniaminov
Data Scientist / CEO
HS Analysis GmbH

Sergey Biniaminov – HS Analysis GmbH und Prof. Dr.-Ing. Sascha Seifert – Medizinische Informatik und Bildverarbeitung HS Pforzheim (v.l.n.r.)

Viktor Eberhardt – HS Analysis GmbH Software-Projektleitung
Der Hochschulprofessor Matthias Wölfel aus Karlsruhe verband in seinem Vortrag die Themen Perzeption und Intuition mit dem Design in der Softwareentwicklung und neue Kommunikationskanäle zwischen Mensch und Maschine. So kann das Verhalten vom Nutzer nicht primär auf die Interaktion mit der Maschine ausgelegt sein. Die KI interpretiert das Verhalten des Nutzers und löst selbständig Handlungen der Maschine aus.

Prof. Dr.-Ing. Matthias Wölfel – Professor für Intuitive und Perzeptive Benutzungsschnittstellen HS Karlsruhe
Simon Sulzer (Digital Hub Initiative), Christian Wiegand (Pioniergarage) und Ludger Pfanz (HfG Karlsruhe) stellten sich den Fragen des Moderators, Michael M. Roth (Team HS Analysis), zum Thema Ethik und Künstliche Intelligenz. Thematisiert und diskutiert wurde der Begriff der Ethischen Intelligenz, der in einem HS Analysis Blog von Michael M. Roth 2018 erschienen ist. Schon 2001 hat der deutsche Philisoph Karl-H. Brodbeck einen Artikel „Zur Ethik der Intelligenz“ resümiert: „Ethik sei nicht zwingend ein Teil der Intelligenz„. Die Scion Teilnehmer haben sich gefragt: „Könnte die Ethik nicht zu einem verbindlichen Element von Mensch- Maschine-Kollaboration werden und was müssen wir dafür tun? In der Diskussion wurde das 1. intelligente Dialogprogramm namens Eliza, das im Jahr 1966 von Joseph Weizenbaum erstellt wurde thematisiert und auch aus dem Jahr 1965 das Moorsche Gesetz, nach dem sich die Komplexität von integrierten Schaltkreisen ca. alle 18 Monate verdoppelt. Darauf aufbauend, fragten sich die Teilnehmer, warum sind wir ausgerechnet heute im Jahr 2018 der KI oder gar einer Superintelligenz näher denn je zuvor und welche Attribute oder Paradigmen dürfen beim akademischen Denken und unternehmerischen Handeln nicht fehlen? Würden wir Menschen uns selbst für vertrauenswürdig in den Augen einer zukünftigen KI halten?

Podiumsdiskussion mit Michael M. Roth (Kommunikation HS Analysis GmbH), Ludger Pfanz (Dozent HFG Karlsruhe), Christian Wiegand (Vorstandsvorsitzender Pioniergarage), Simon Sulzer (Projektleiter de:hub Karlsruhe) – v.l.n.r.
Parallel zum Forum (Vorträge, Podiumsdiskussionen, Workshops) fand ein Hackathon statt, der sich unter den Studierenden mit interdisziplinärem Interesse (Fachbereiche Informatik, Biologie, Medizin, Design) einer zunehmenden Beliebtheit erfreut.

Hackathonteilnehmende Scion 2018 an der Challenge von Bayer AG

Scion Hackathon 2018

Scion Hackathon 2018 – Gruppe Zeiss

Scion 2018 Hackathongruppe

Scion 2018 Hackathongruppe Karlsruhe

Oguzhan Angay von Zeiss als Betreuer von Scion Hackathon 2018
Hier wurde beispielsweise eine komplette App zur Diagnose von Hautkrebs mit Deep Learning nur an einem Wochenende entwickelt. Das Team hat ein aufbereitetes Ground Truth Dataset von Hautkrebsarten bekommen. Nach dem verschiedene Netzarchitekturen verglichen worden sind, hat sich das Team für das ResNet Inception v2 entschieden. Es wurde 20% drop out verwendet und pretrained antrainiert, d.h. etwas Vortrainiertes auf dem ImageNet geladen und anschließend darauf trainiert. Kombiniert mit den residuals, drop outs vom ResNet wurden an der Stelle auch die inception blocks, die sehr gut verschiedene Merkmale an verschiedenen Skalen extrahieren können, verwendet. Um möglich beste Ergebnissicherheit zu haben, hat das Team Ensembles verwenden, z.B. gleiche Bilder wurden durch das ResNet 50 und das ResNet Inception v2 analysiert, Ergebnisse grafisch dargestellt und letztendlich die Entscheidung für das best performende Netz getroffen.
Auch nur an einem Wochenende hat ein anderes Team ein Deep Learning Modell für die Detektion verschiedener Strukturen in den WSI Mikroskopiedaten entwickelt. Dabei wurde ein Fully Convolutional Network (FCN) auf Basis der Architektur VGG 16 Net, welches auf ImageNet vortrainiert und von Keras erweitert war, mit ca 4.000 Bildern und Masken auf Google Colab antrainiert. Das Ziel dieser Challenge war es, die Ärzte durch KI in der Diagnostik zu unterstützen.
Eine weitere Gruppe erarbeitete einen Algorithmus für die Quantifizierung der Gewebeschnitte in der Medikamentenentwicklung. Es ist eine Softwarelösung entstanden, die den maximalen Abstand von Tumorzellen von den Target positiven Zellen an humanen Gewebeschnitten ermittelt. Die Tumorzellen sollen anhand der Morphologie der Zellen nach einem Trainingsset erkannt werden. Weiterführend kann die Tumor vs. Stroma Erkennung mittels Training Set entstehen.
Auch bildgebende Challenges für die Mikroskopieplattform APEER wurden an diesem Wochenende von Hackathon-Gruppen gelöst und auf APEER hochgeladen. Insgesamt waren 10 Gruppen mit verschiedenen Aufgaben an dem Wochenende beschäftigt. Hier gibt es die komplette Aufgabenliste beim Hackathon.

Richard, Nadim und Xizhe – Pitch der Hackathongruppe Medikamentenentwicklung
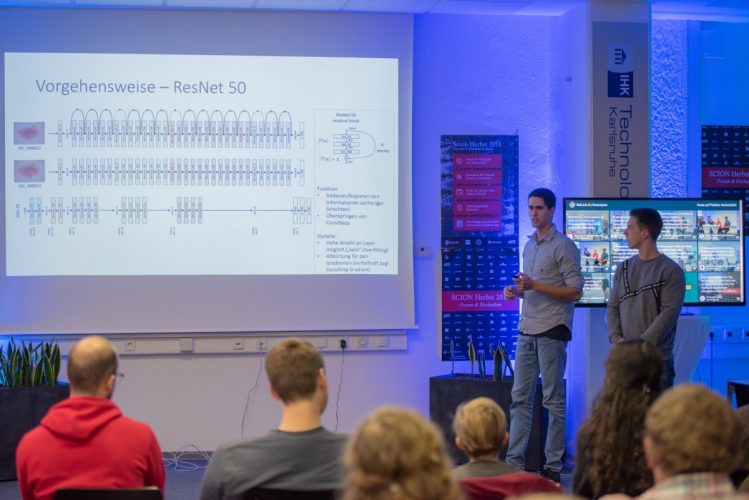
Michael, Sven – Pitch Hackathongruppe AI in der Diagnose
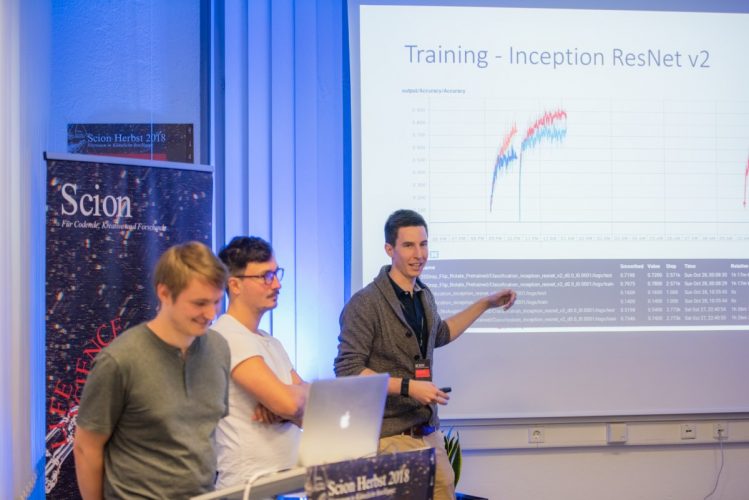
Matthias, Sebastian, Julian – Pitch Hackathongruppe App in der Diagnose

Max, Timo und Katharina – Pitch Hackathongruppe Strukturdetektion in WSI

Florian und Karol – Pitch Hackathon APEER Gruppe
Jeder Teilnehmer durfte auch persönlich ein Zeichen gegen Krebst setzen, sich typisieren lassen und somit letztendlich die Stammzellen an die Deutsche Stammzellspenderdatei vor Ort spenden.

Daniela und Frank Stoetzer – Stammzellspenderdatei
Bei Scion Herbst 2018 ist es uns im Vergleich zu Scion Sommer 2018 gelungen, die Zahl der Teilnehmenden deutlich zu erhöhen und mehr Interessenten sowohl für die inhaltlichen Fragen von Life Science und Künstlicher Intelligenz als auch für das Veranstaltungsformat Scion selbst zu gewinnen. In 2019 planen wir die Fortsetzung der Veranstaltungsreihe und freuen uns abermals auf eine Vielzahl von Interessenten, spannende Themen und interessante Begegnungen.

Scion Herbst 2018
Last but not least: Eine Nennung unserer Sponsoren und Partner im Detail, die sich alle sehr darum bemüht haben, Scion 2018 zum Erfolg zu führen. Herzlichen Dank nochmals dafür an Euch: Zeiss Microscopy und die Zeiss-Plattform APEER, Bayer Pharma AG, Karlsruhe Institut für Technologie, UKE Hamburg, CyberForum, baden württemberg: connected (bwcon), Technologiefabrik Karlsruhe, PionierGarage Karlsruhe, BioLAGO, BioRN, KI Bundesverband e.V., Pathologie Molekularpathologie Pforzheim (PMP), Digital Hub Initiative, BEYOND Festival Future Design, karlsruhe.digital, eit Health, BIO PRO Baden-Württemberg, Walls.io, Hochschule Technik Wirtschaft Karlsruhe, Hochschule Pforzheim, Universitätsklinikum Mannheim – Pathologisches Institut, Universitätsklinikum Freiburg, Duale Hochschule Baden-Württemberg Karlsruhe, Hochschule für Gestaltung Karlsruhe, Städtisches Klinikum Karlsruhe, Deutsche Stammzellspenderdatei und ein ganz besonderer Dank geht an Jürgen Siegloch für die Begleitung und Koordination von Scion 2018 in allen Planungsphasen.
Für weitere Informationen zu Scion http://scion.hs-analysis.com/
Rückblick: Scion Herbst 2018 “Vertrauen in KI” Dezember 1st, 2019hsa-admin